ハイライト
生物が新しいアミノ酸を獲得するしくみ
2010年11月25日
生物の体の大部分はアミノ酸がつながってできたタンパク質からできています。タンパク質は20種類のアミノ酸の組み合わせによって作られ、ヒトの体内で作られているタンパク質は10万程もあるといわれています。タンパク質の種類の多さと複雑さは、その部品であるアミノ酸の種類が多くあるためです。生物は最初から今のように20種類のアミノ酸を持っていたのではなく、進化とともに徐々にアミノ酸の種類を増やし、より複雑なタンパク質を作り出していったと考えられています。
今回紹介する研究は、普通とはちょっと違う方法でアミノ酸を作り出している酵素のお話です。わたしたち真核生物より原始的な生物の真正細菌が持つこの方法は、生物が新しい種類のアミノ酸をどのように獲得してきたのか、進化の一コマまで垣間見せてくれます。
DNAからタンパク質を作る
生物の設計図であるDNAには、どんなタンパク質を作るかという情報が書かれています。タンパク質が作られるときには、必要に応じてほどけたDNAを鋳型にしてG,C,C,A,U…と情報のコピーが作られます。このコピーが伝令RNA(mRNA)で、文字列の3文字を1組(コドン)にして、それが1つのアミノ酸へと変換されます。こうして次々と変換されたアミノ酸が鎖のように連なり、タンパク質になります。
文字列からアミノ酸へ変換する時、アダプターのように働くのが転移RNA(tRNA)です。tRNAの中には3文字のコドンと対になるアンチコドンがあり、この2つが正しくペアになることで、コドンの情報がtRNAに読みとられます。一方、アミノ酸はそれぞれに対応するtRNAにくっつき、アミノアシルtRNAを作ります。
ここでアミノ酸のtRNAへの付加が正しいペアで起こることが、正確なタンパク質合成の大前提となります。この反応を担うアミノアシルtRNA合成酵素は20種類のアミノ酸それぞれに専用のものが用意されていて、アミノ酸とtRNAが正しいペアを組むようになっています。
しかし、多くの真正細菌にはアミノ酸の一種グルタミン(Gln)をtRNAに付加するアミノアシルtRNA合成酵素であるGlnRSが存在しません。もちろん真正細菌の遺伝子にもグルタミンに対応する情報が書かれていますし、タンパク質の部品としてグルタミンが使われているのにもかかわらずです。彼らは一体どのようにしてグルタミンをタンパク質の部品にしているのでしょうか。
似たアミノ酸で代用し、あとから修正
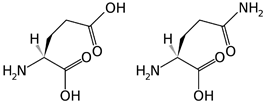
20種類のアミノ酸の中には、グルタミンによく似た「グルタミン酸(Glu)」があります。グルタミン酸の-OH(ヒドロキシル基)が-NH2(アミノ基)になっているのがグルタミンです(図1)。GlnRSのない真正細菌では、グルタミン用のアダプターtRNAGlnにも、グルタミン酸用のアダプターtRNAGluにも、どちらにもグルタミン酸(Glu)が付加されることが分かっていました。正しいペアを付加する酵素が無いので、似ているグルタミン酸を代わりにくっつけてしまおう、といった模様です。しかし似ているとは言っても間違えているペアですから、「アミノ酸とtRNAが正しくペアを組む」という正しいタンパク質合成の大前提が崩れてしまっています。
正しいタンパク質を作るにはどうしているのでしょうか。誤ったペアGlu-tRNAGlnにはGatCABという酵素が働いて、グルタミン酸 (Glu) の-OHを-NH2に変換しているのです。これは、わざと間違えたペアを作り、後から修正するという、少し変わったやり方です。
初めて見えた巨大分子
GatCABはどうやって誤ったペアを見分けて、修正を行なっているのでしょうか。東京大学の横山茂之(よこやま・しげゆき)兼任教授と伊藤拓宏(いとう・たくひろ)特任助教らの研究チームは、まず真正細菌から取り出したグルタミン用のtRNAGln、グルタミン酸用の(誤ったペアを作る)アミノアシルtRNA合成酵素GluRS、そしてアミノ基への変換を行なう酵素GatCABの三者の結合状態を調べました。その結果三者は安定した巨大な複合体を作ることがわかり、これを「グルタミン・トランスアミドソーム」(グルタミンを作り出すためにグルタミン酸にアミノ基を転移する複合体という意味)と名付けました。
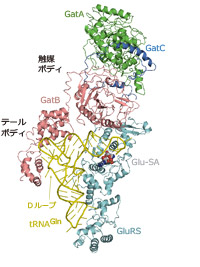
次に研究チームはこの巨大な複合体の結晶を作ることに成功し、KEKフォトンファクトリーのAR-NE3AとSPring-8の放射光を用いたX線結晶解析装置で、その立体構造を調べました(図2)。
図2
画像提供:東京大学大学院理学系研究科・理化学研究所生命分子システム基盤研究領域
グルタミン・トランスアミドソームの立体構造。tRNAGln(黄色)にGluRS(水色)とGatCAB(緑、青、ピンク)が同時に結合している。GatCABは、そのテールボディにおいて、tRNAGlnを認識している。tRNAGlnの末端部分に、原子をボールで表しているのがグルタミン。一方で、触媒ボディ(実際にアミノ基への変換を行なう部分)はtRNAGlnに接してはいなかったが、その一部は、GluRSと相互作用し、「留め金」がかかったように固定されている。これにより、GatCABがGluRSによって生成される中間体Glu-tRNAGlnを受け渡されるのを待ち構えていることが判明した。
グルタミン用tRNAGln(黄)にグルタミン酸用の酵素GluRS(水色)がくっついている時、これは誤った組み合わせなので、すぐに修正する必要があります。修正する酵素GatCABの一部であるGatB(ピンク)は、tRNAのL字型の構造の外側の部分を見分けて、修正の必要なtRNAGlnであるか、修正不要のtRNAGluであるかを判断していることがわかりました。
しかし、修正されるべきグルタミン酸は、tRNAのL字型の末端部分に付いていて、修正する酵素のGatCABとは遠いところにあるように見えます。いったいどのようにして、GatCABは、tRNAの末端にあるグルタミン酸にアクセスしているのでしょうか。
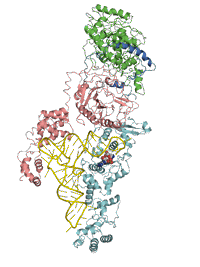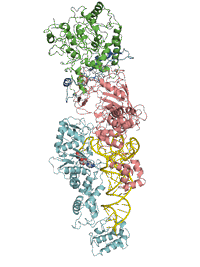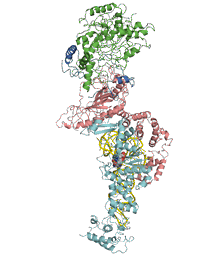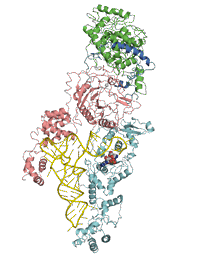
他の生物種のGatBや以前にフォトンファクトリーのAR-NW12Aで構造解析した単体のGluRSの構造と比較すると、どちらにも蝶つがいのような構造があることが分かりました(図3)。すなわち、2つの酵素は、tRNAに結合したまま大きく折れ曲がることで、グルタミン酸が結合している部分にアクセスできるようになっていたのです(図4)。
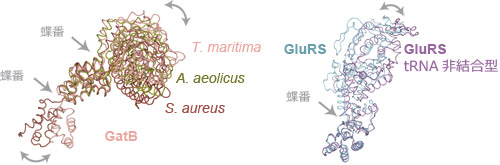
図3
写真提供:東京大学大学院理学系研究科・理化学研究所生命分子システム基盤研究領域
左図:3種類の生物種のGatBの構造を重ね合わせたところ。共通して2個所(矢印)に蝶つがいのような構造があることがわかった。
右図:tRNAに結合していないGluRSと、トランスアミドソーム中のGluRSの構造を重ね合わせたところ。1個所(矢印)つがい構造があることがわかった。この構造により、GatCABとGluRSは、tRNAGlnに結合したまま(トランスアミドソームを形成したまま)で、触媒ボディを動かすことが可能となる。
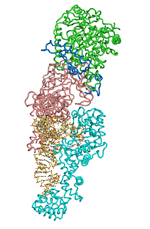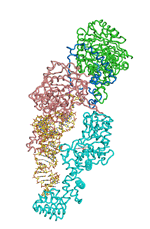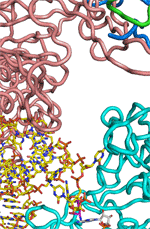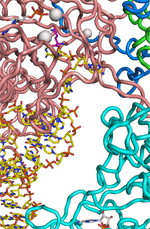
図4
画像提供:東京大学大学院理学系研究科・理化学研究所生命分子システム基盤研究領域
グルタミン‐トランスアミドソームにおける中間体Glu-tRNAGlnの受け渡し機構。
(A) GluRSが間違ったペアであるGlu-tRNAGlnを生成する。(B) 次にGluRSは、蝶つがい構造を使って、アクセプターアームと呼ばれるグルタミン酸 (Glu) の付加されたtRNAの末端部分から離れる。(C) その後、待機しているGatCABが、自身の触媒ボディをtRNAの末端部分に近づけて、グルタミン酸が結合している部分を受け取り、グルタミン酸をグルタミン (Gln) へと修正する。
ムービーを見るにはQuickTime Playerのプラグインが必要です
![]() ダウンロードはこちら
ダウンロードはこちら
今回の研究でわかったしくみは、生命が新しいアミノ酸をタンパク質の部品として獲得するひとつの戦略を見せてくれています。新しいアミノ酸を取り入れることは、タンパク質のバリエーションが増えることにつながりますが、同時に誤ったタンパク質を作ってしまう大きなリスクも抱えています。
「生物がリスクを冒しながらも、新たなアミノ酸を獲得してきた、ということは研究者の間では知られていました。でも、生物が生き残るにはそんなに無謀なことはしない、きっとリスクを軽減するしくみがあるはずだと思っていました」そう語っていた伊藤さんにとっても、この蝶つがいの巧妙な動きは予想外だったようです。
この研究は「ターゲットタンパク研究プログラム」、および「日本学術振興会 科学研究費補助金 基盤研究(A)」の一環として行われたもので、英国の科学雑誌「Nature」の9月29日版に掲載されました。
![]() 毎週の記事のご案内をメールマガジン「news-at-kek」で配信します。
毎週の記事のご案内をメールマガジン「news-at-kek」で配信します。
詳しくは こちら をご覧ください。