北海道大学 大学院薬学研究院の前仲 勝実 教授、大阪大学 免疫学フロンティア研究センター/微生物病研究所の荒瀬 尚 教授らの研究グループは、単純ヘルペスウイルスが宿主の免疫細胞から排除されることなく、体内へ入り込む巧妙な感染機構をフォトンファクトリー(PF)、SPring-8を利用したタンパク質の立体構造の分析から原子レベルで解明しました。
単純ヘルペスウイルスは脳炎や口唇ヘルペス、性器ヘルペス、皮膚疾患、眼疾患、小児ヘルペスなど、多様な疾患を引き起こす病原性ウイルスで、日本でも年間約7万人が治療を受けていると言われています。特に、性器ヘルペスは既存の抗ウイルス薬では完治が不可能であること、ヘルペス脳炎では致死的もしくは重度の後遺症が残る場合もあります。単純ヘルペスウイルスの感染機構の解明は、感染予防や治療のために重要な情報となります。
これまでの研究で、単純ヘルペスウイルスが細胞へ侵入する際には、ウイルスが免疫細胞表面にあるPILRαタンパク質と結合することで、免疫細胞の攻撃を抑え、感染するということが分かっていました。しかし、それらの結合の構造的なしくみは解明されていませんでした。
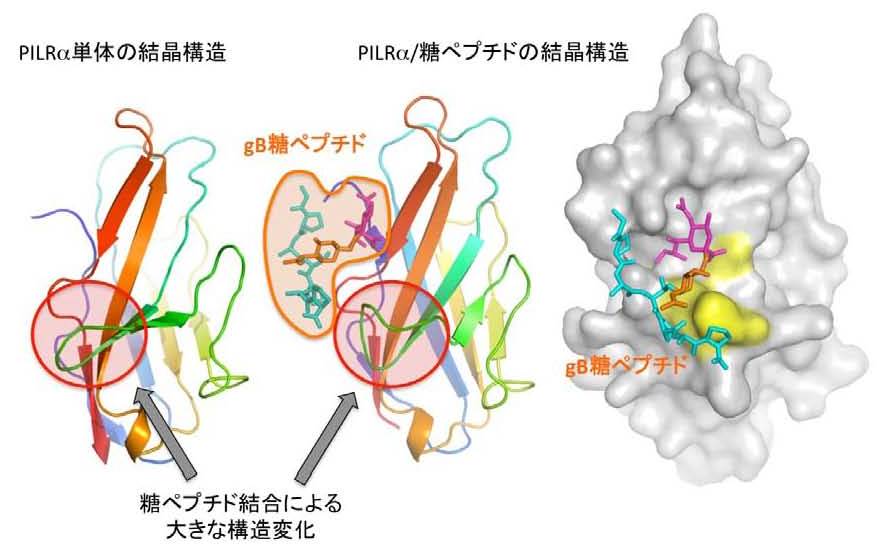
今回、研究グループはウイルスと結合する前のPILRα単体、およびウイルス表面にある糖タンパク質と結合した状態のPILRα複合体の構造をX線回折実験によって決定しました。 その結果、PILRαはウイルス表面にあるgBペプチドを含むシアル化O型糖鎖※を認識することが分かり、単純ヘルペスウイルスが侵入する際に、このアミノ酸からなる糖ペプチドを添加すると、PILRαのgB結合部位がふさがれ、侵入阻害剤として利用できることが分かりました。
この研究はJST戦略的創造研究推進事業 チーム型研究(CREST)「アレルギー疾患・自己免疫疾患などの発症機構と治療技術」の一環として行われました。本成果は、米国科学雑誌「米国科学アカデミー紀要(PNAS)」のオンライン速報版に2014年6月2日で公開されました。
北海道大学 プレスリリース>>単純ヘルペスウイルスが宿主に感染するメカニズムを解明
シアル酸を含むO型糖鎖。O型糖鎖は、タンパク質のセリンまたはスレオニン残基に結合している糖鎖のこと。