東京大学大学院薬学系研究科の清水敏之教授、大戸梅治講師、大阪大学大学院工学研究科の内山進准教授らの研究グループは、微生物の侵入を感知して自然免疫応答を活性化するTLR9受容体の詳細な立体構造を、フォトンファクトリーAR-NE3AおよびSPring-8を用いて世界で初めて明らかにしました。
細菌やウイルスなどの病原体への感染を防ぐ仕組みとして、私たちの体には自然免疫機構が備わっており、Toll様受容体(TLR)と呼ばれるタンパク質が主要な役割を担っています。TLRは、病原体のもつ分子によって活性化され、二量体を形成することでその役割を果たすことが知られています。今回立体構造を明らかにしたTLR受容体の一種TLR9は、微生物由来のDNA配列(CpGモチーフ(注1)を感知することで、インターフェロン(注2)などの産生を促すことが知られていますが、具体的にどのようにこのDNA配列を認識するのかは不明でした。
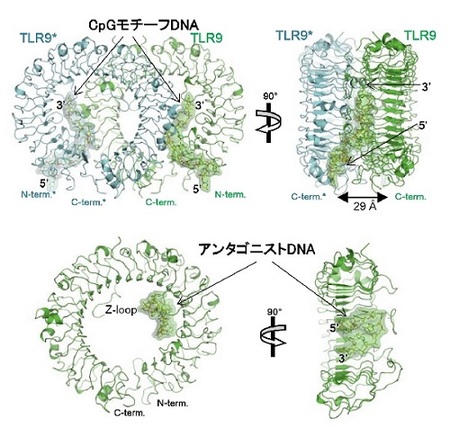
図1:TLR9の結合様式
(上)CpGモチーフを有するDNA配列とTLR9との結合様式。 (下)アンタゴニストDNA配列とTLR9との結合様式。
2量体を形成するTLR9分子の一方を緑色で、他方を青色で示している。CpG モチーフは伸びた構造で、TLR9の2量体に2ヶ所で結合している(2 対2複合体)。
アンタゴニストDNAはTLR9の馬蹄型構造の内側にループ構造を作り結合している(1 対1複合体)。
研究グループは、DNA配列が結合していないTLR9、CpGモチーフを有するDNA配列が結合しているTLR9、TLR9の機能を阻害するDNA配列(アンタゴニストDNA配列)が結合しているTLR9の3種の立体構造を明らかにしました。その結果、TLR9とCpGモチーフは2対2の比率で結合し二量体の活性化型を形成することが分かりました(図1上)。このDNA配列はTLR9のN末端側にある溝に結合することによって認識されていることが分かりました。一方で、TLR9とアンタゴニストDNA配列は1対1の比率で結合し、二量体になることはありませんでした。また、このDNA配列はTLR9の馬蹄型構造の内側にコンパクトなループのような形で結合していました(図1下)。
TLR9は、抗ウイルス薬やアレルギー薬、ワクチンなどの創薬の標的として注目されています。今回の研究成果によりTLR9を活性化・不活性化するDNA配列との結合様式が明らかになったことで、これら治療薬の設計につながるものと期待されます。
本成果は英国の科学誌Natureのオンライン版に2015年2月9日付で掲載されました。
東京大学発表のプレスリリースはこちら
論文情報
Structural basis of CpG and inhibitory DNA recognition by Toll-like receptor 9
Umeharu Ohto, Takuma Shibata, Hiromi Tanji, Hanako Ishida, Elena Krayukhina, Susumu Uchiyama, Kensuke Miyake and Toshiyuki Shimizu
Nature (2015) doi:10.1038/nature14138
DNA中に存在するシトシンとグアニンがホスホジエステル結合でつながったDNA配列。哺乳類ではメチル化されることが多いのに対して、細菌やウイルスではメチル化されていない(非メチル化)ことが知られている。非メチル化CpGモチーフはTLR9を強く活性化してさまざまな免疫応答を引き起こす。
細菌やウイルスなどの病原体の侵入に対して免疫系の細胞が分泌するタンパク質で、ウイルスの増殖を抑制する作用や免疫系を活性化するなどの作用を発揮する。