物構研は、放射光・中性子・ミュオン・低速陽電子という4つの量子ビームの実験施設を運用し、それを活用した研究を行っています。 近年、生命科学分野での必要性から、新しい量子ビーム「電子線」が加わりました。 構造生物学研究センター(SBRC)に導入された透過型電子顕微鏡(TEM)です。 TEMは、ちょうど昔のテレビに用いられていたブラウン管のように電圧によって加速した電子を試料に当て、透過した電子から試料の透過像や回折像などの情報を得る装置です。 生命科学分野では、試料を低温に冷やしたまま観察できるクライオ電子顕微鏡と呼ばれるTEMが用いられます。その画像データを「単粒子解析」という特殊な手法で解析することで、タンパク質粒子の立体構造を知ることができます。
物構研の構造生物学研究センター(SBRC)では、生命の謎や病気のしくみなどの解明のため、生命体の主要な構成要素であるタンパク質を調べています。タンパク質を構成するアミノ酸は基本20種類ですが、その組み合わせによって多様なタンパク質が存在し、それぞれ複雑な立体構造を持ちます。タンパク質の立体構造が分かると、そのタンパク質がどう動くのかが分かり、どんな働きをするのかを知ることができます。だから、構造生物学者たちはタンパク質のかたちを知りたいと考えるのです。
分子構造の長さを表すときには慣例としてÅ(オングストローム)という単位を使います。例えば、水分子の酸素ー水素結合の距離はおよそ1Åです。SI単位系では1Å=10-10m=0.1 nmです。タンパク質は小さなもので10Å程度、大きなものでは1 ㎛以上もあるそうです。
タンパク質の立体構造を知る方法として、同じタンパク質が規則正しく並んだ状態(結晶)にして放射光(X線)を当て、結晶による反射が強めあったり打ち消しあったりする情報(回折(かいせつ)像)を検出し解析する方法があります。フォトンファクトリー(PF)では、このX線結晶構造解析によって数々のタンパク質の構造が調べられています。
しかし、タンパク質を結晶にするのは難しく、試行錯誤を重ねてもなかなか結晶にならない例も少なくありません。そこで開発されたのが、結晶化の必要がない「クライオ電子顕微鏡による単粒子解析」で、2017年、研究開発に貢献した3名にノーベル化学賞が授与されました。
透過型電子顕微鏡(TEM)は、数百kVの電圧で加速した電子を試料に当て、試料を透かして見たときの濃淡のある影絵のような像(透過像)を得る顕微鏡です。虹色に分かれる可視光で撮った写真ではないので、TEM写真には色の情報はありません。しかしその分解能*は、条件が合えば原子の粒が見えるほど高いのです。
*分解能:対象を識別できる能力のこと。例えば、人間の肉眼の分解能は0.1 ㎜程度。TEMの分解能は数Å程度。
量子力学の創成期、ド・ブロイが提唱した物質波の考えによれば、電子も波と考えることができます。波としての電子の波長を計算してみると、加速電圧が200 kVのときおよそ2.7×10-12 m=0.027Åで、可視光の波長(3×10-7~8×10-7 m)に比べてはるかに短いことが分かります。波長が短い波ほど小さいものを識別できるので、Åで測るような細かい構造を知るために電子線は有効なのです。参考までに、PFでよく使われる放射光の波長は、10-10~10-7m(1Å~100 nm)程度です。
ただしX線と違って、電子線は透過力が弱いので、試料はとても薄くなければならないだけでなく、電子線の通り道も空気中の分子をできるだけ除いた状態(真空)にする必要があります。そのような環境にタンパク質を入れたらどうなるでしょう。真空であることで元の形を保っていられず、干からびてしまうでしょう。また電子線が当たることで化学反応が起こり、タンパク質は壊れていきます。それでは知りたかった元のかたちの観察はできません。そこでタンパク質を極低温の薄い氷に閉じ込めて観察する方法が考案されました。この氷は、水分子が規則正しく並んだ結晶ではなく、不均一なガラスのような構造になっています。このガラス状の氷によってタンパク質は真空から守られ、また低温であるほど化学反応が進みにくくダメージが少ないと言われています。
試料とその周辺を液体窒素で-200℃くらいの低温に冷やしながら観察できるTEMを、クライオ電子顕微鏡、略してクライオ電顕と呼びます。SBRCでは2018年の初めに導入しました。
 試料グリッドを作成しているところ
試料グリッドを作成しているところ クライオ電顕の試料ホルダー周辺
クライオ電顕の試料ホルダー周辺 自動撮影をしながらデータ処理をしているところ
自動撮影をしながらデータ処理をしているところQ. 単粒子解析用のクライオ電顕と、一般の低温で観察できる電顕は何が違うの?
A. 単粒子解析では形態観察に比べて桁違いの枚数の撮影をします。単粒子解析に使うクライオ電顕は、撮影の自動化が進んでいることが大きな違いです。

単粒子解析は、その名の通り、ひとつひとつばらばらの粒になったタンパク質のTEM写真から、その立体構造を知るための方法です。同じタンパク質のたくさんの粒のTEM写真を撮り、その二次元の情報から三次元像を復元します。
多数の二次元像から三次元構造を再構築するという例では、病院などで行うX線CTがあります。X線源あるいは撮影対象を回転させ、少しずつ角度を変えて、1つの立体物のあらゆる角度からのX線写真(投影像)を撮ります。二次元の情報は、コンピュータで三次元に再構築されますが、複数の投影情報の辻褄が合うところの信号が強くなり、合わないところは弱くなって、三次元構造が得られていきます。この場合はあらかじめ投影した角度が分かっているのでその情報を再構築に使うこともできます。
さて、タンパク質のTEM像の場合はどうでしょう。タンパク質の粒は薄い氷に閉じ込められて固定されています。しかも、タンパク質の粒の向きは初めからバラバラ……。!
ここで発想の転換です。1つの粒をあらゆる角度から撮影することができない代わりに、初めからあらゆる方向を向いている(と思われる)たくさんの同じタンパク質の粒を撮影すれば、得られる写真は同じではないでしょうか?
ただ、違うのは、どの角度から撮った写真なのかが分からないこと。つながりの分からないバラバラの写真だけが多数得られるということです。
ちょっとパズルのような話になってきました。バラバラの写真の山から推理して元のストーリーに組み立てる、そんな探偵のような作業が必要なのです。
この探偵業の難しいところはもうひとつあって、元の写真がくっきりしていないこと。タンパク質のダメージを防ぐため弱い電子線を当てているからです。例えて言うなら、ろうそくの灯りで撮った写真のようなもの。解析にはノイズの多いざらざらした画像を使わざるを得ないのです。
 単粒子解析に用いられるTEM写真の例
単粒子解析に用いられるTEM写真の例
 「守屋です。私の単粒子解析の相棒はこのコンピュータです。」
「守屋です。私の単粒子解析の相棒はこのコンピュータです。」
ここからは単粒子解析を行う探偵のような研究者、自称「画像処理屋」の守屋 俊夫 特任准教授の手法をご紹介しましょう。
まず、撮影したTEM写真から粒子の部分を切り出します。粒子の大きさにもよりますが、1枚のTEM写真には100~200個ほどのタンパク質の粒が写っています。粒同士が距離を保って散らばるように試料を作成しているので、一定の余白をとって1粒ずつ1枚の画像に切り出すことができます。ここで活躍するのが粒子ピッカーと呼ばれるソフトウェアで、ディープラーニング(深層学習)を使って自動で粒子を認識し拾い上げを行います。
しかしこの中には、氷の結晶やカーボン膜の縁、壊れたタンパク質のかけらなどが写り込んでいる「ごみ画像」も入っています。ピッカーが拾い上げた後、基準から外れているものは自動で捨てられていきますが、その基準を与えるのは人です。基準を厳しくしすぎると、大切なタンパク質の粒画像まで捨ててしまいますから、ごみとそうでないものの判別をどう機械に任せるか、これも腕の見せどころ。こうしておよそ2000枚のTEM写真から数十万から百万の「原画像」が得られます。
データにノイズはつきもの。このノイズを取り除くには、同じようなデータをたくさん重ねるのが王道です。ノイズは真の情報ではないので、ランダムな場所にランダムな強さで現れます。だから、たくさんのノイズを重ね合わせれば、打ち消し合って弱くなる。これがノイズ除去の考え方です。
重ね合わせるためには、同じ投影像は、原画像中で同じ角度・同じ位置になければなりません。回転と位置合わせの作業が必要です。それによって二次元画像を大まかに合わせて、投影方向ごとに分けることを「二次元クラス分け」と呼んでいます。
さて、回転・位置合わせと言っても、何か基準がないと合わせられませんね。こんな投影像になるはずと基準を与える方法Aもありますが、何もない状態からクラス分けをさせる方法Bもあります。方法Bでは、原画像から適当に数枚を選んで「この像に似たもの集まれ~」と、何度も位置合わせや回転をかけて、形が似ているものを集めます。基準のかたちに近いものは同じクラス、遠いものは違うクラスと原画像を振り分けていくと、似た画像が集まったクラスが数百できます。クラスごとに平均像を計算すると、ノイズが少ない像ができるので、これを基準にして次の作業に移ります。
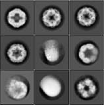 クラス平均像の例
クラス平均像の例
しかし、この時点でもごみが残っています。計算は自動で行っても、どの絵がきれいか、ごみクラスができていないか、最終的に選ぶのは人の目ということになります。この過程で数百枚のクラス平均像ができます。
 選定後のクラス平均像の例
選定後のクラス平均像の例
さて、いよいよ三次元の大まかな構造を作り上げるのですが、それにもいろいろな手法があります。
上記ⅲの方法で、どの角度から見ても矛盾のない初期三次元構造ができあがりました。 次は、統計手法を使って三次元角度と位置合わせを最適化します。
 初期三次元構造の例
初期三次元構造の例
できあがった複数の三次元構造と、原画像とのプロジェクションマッチングを行ないます。プロジェクションマッチングを繰り返し、「この原画像は、どの立体構造のどの角度との類似度が最も高いか」を探して、原画像を立体構造ごとに分類していきます。
立体構造ごとに分けた原画像のグループの中で、より精密な構造を再構築していきます。3.では、同じタンパク質でも構造が違うものが混ざっている(かもしれない)状態で三次元再構築をしましたが、今度は同じ構造を持つと思われる原画像だけを抜き出した状態での再構築です。
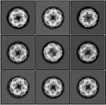
 三次元精密化前の像の例
三次元精密化前の像の例
再度プロジェクションマッチングをして類似度が高いものを集めて三次元再構築する…。この最適化を25回くらい繰り返すうちに、だんだん推定した三次元角度が正確になっていき三次元像がきれいになってきます。いいデータセットの場合は、数日の計算で3Åくらいの分解能で構造を解析できます。
 三次元精密化後の像の例
三次元精密化後の像の例
関連記事:最適化について ―どこがもっとも類似度が高いのか―
【解析結果の例】:
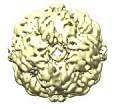
SBRCでの単粒子解析による酸化還元酵素「硫黄オキシゲナーゼ/レダクターゼ」
協力:東京大学大学院 農学生命科学研究科 伏信 進矢 教授
赤や黄色に見える部分は突起しているためチムニー(煙突)と呼ばれ、硫黄の出入り口ではないかと考えられている。色は分解能の目安で、青いほど構造の揺れが小さいために分解能が高く、赤いほど構造の揺れが大きい可能性が高いことを示す。東京大学との共同研究によるこのタンパク質の解析では、全体の平均で2.05Åという高い分解能が得られた。
Q. クライオ電顕で撮影をしたら全てのデータを単粒子解析するの?
A. いまSBRCではスクリーニングで500枚程度撮り、そこでいい感触が得られれば本撮影を行います。本撮影では2000枚程度のTEM写真を撮り、それが単粒子解析の対象となります。
初期三次元再構築や三次元クラス分けで、構造が複数あるとするのはどうしてでしょう。試料の中に別のタンパク質やごみが混ざっている場合の受け皿として? いえ、それだけではありません。
タンパク質は本来、複数の異なる構造を持つものです。タンパク質は生命体の中で様々な働きをしますが、文字通り動いているので、その構造も変化するのです。もしクライオ電顕による単粒子解析で同じタンパク質の複数の構造が捉えられたとしたら、それはタンパク質の粒が氷に閉じ込められる瞬間の構造の違いを捉えたということです。
タンパク質が、例えばαという構造からβという構造に姿を変えるとします。αからβへの過渡期にはαともβとも違う構造があるはずですが、タンパク質は安定した位置まで短時間に一気に動く性質があるので、途中の状態で固定される可能性はとても小さいと考えられます。また、タンパク質の外側にはループと呼ばれる柔らかい構造がありますが、ループは動きやすいことが多いため平均化によって相殺されて見えないことがよくあります。
1つのタンパク質結晶から得た結晶構造解析データでは複数の構造を捉えることはできないので、複数の構造が見えることは単粒子解析の利点のひとつとされています。
単粒子解析の分解能は、通常の分解能の定義とは違い、FSC(Fourier Shell Correlation、フーリエシェル相関)分解能と呼ばれ、「特定のデータセットを2つに分けて、それぞれ別々に処理してもその分解能(周波数の逆数)の情報までは、いつも一致して再構築できる」という指標です。
また、単粒子解析では、分解能は構造の局所ごとに異なります。表紙のタンパク質の構造図は、分解能によって色分けをしたもので、基本的に、よく動く場所ほど分解能が低く、構造が安定している場所は高く表示されています。
単粒子解析の世界最高分解能は1.15Åです。SBRCでは平均2.5~4.0 Å程度、最高2.05Åです。
SBRCのクライオ電顕による単粒子解析は共同利用の軌道に乗ったばかり。大学や企業などから新たなユーザーが続々と訪れています。経験と知見を積み重ね、分解能向上はこれから大いに期待できます。
 守屋氏が主宰する単粒子解析の研究会のようす
2020年10月 KEKつくばキャンパス COI棟にて
守屋氏が主宰する単粒子解析の研究会のようす
2020年10月 KEKつくばキャンパス COI棟にて
タンパク質の二次元の電顕像から三次元の構造を再構築する手法の研究をしている。アルゴリズムを開発するには全体の流れを把握する必要があり解析実務も担当。
学生時代は神経学や認知神経心理学を専攻し、人工知能の研究をしていた。卒業後、電子顕微鏡の開発会社で画像解析のソフトウェア開発をしているときに単粒子解析に出会う。前職のマックスプランク研究所では解析ソフトウェアの開発に従事。KEK着任はクライオ電顕の立ち上げ期間にあたる2018年。クライオ電顕のユーザー受入れが始まってからは、実習型のセミナーなどデータ解析の指導にも力を入れている。パズルが大好き。
 KEKつくばキャンパスCOI棟 クライオ電子顕微鏡前にて
KEKつくばキャンパスCOI棟 クライオ電子顕微鏡前にて
物構研 構造生物学研究センター(SBRC)は、日本国内の研究者がクライオ電顕を利用して高分解能構造解析を行うための「BINDSプロジェクト クライオ電顕ネットワーク」において中心的な役割を果たしています。守屋准教授が講師を務める講習会も開催しています。
関連ページ:
関連記事:
物構研トピックス 2018/04/18 構造生物学研究センターにクライオ電子顕微鏡が導入されました